La confirmation ou l'identification des souches de Legionella est réalisée :
- sur les caractères morphologiques et phénotypiques des colonies (aspect des colonies à la loupe binoculaire, caractères culturaux...) ;
- par immunofluorescence directe à l'aide d'anticorps monoclonaux de Dresden (mise à disposition par Christian Lück, CNR des Légionelles, Dresden, Allemagne) ou grâce aux immun-sérums polyclonaux de lapin préparés par le CNR pour toutes les espèces et sérogroupes de légionelles ;
Lück C, Fry NK, Helbig JH, Jarraud S, Harrison TG. Typing methods for Legionella. Methods Mol. Biol. 2013; 954:119-48.
- par des particules de latex sensibilisées à l'aide de réactifs commercialisés (Oxoid, Prolab) ;
- sur des caractères génotypiques par séquençage du gène mip et comparaison de la séquence à la base de données disponibles sur le site EWGLI (www. ewgli.org) ;
- par spectrométrie de masse MALDI-TOF-MS avec un instrument Vitek-MS (base de connaissance v3.2)
- l'analyse du ST basée sur l'analyse de 7 gènes ménage définis au niveau européen pour le typage des Legionella pneumophila : flaA, pilE, asd, mip, mompS, proA et neuA permettant d'établir un ST en fonction des numéros d'allèle retrouvés.
(Gaia V. et al. Consensus sequence-based scheme for epidemiological typing of clinical and environmental isolates of Legionalla pneumophila. J Clin Microbiol. 2005 May;43(5):2047-52)
C'est l'analyse la moins discriminante. Néanmoins, il peut permettre de différencier des souches pour des clones non endémiques.
- L'analyse de cgMLST (core genome MLST) reposant sur l'analyse de 50 gènes (définis au niveau européen) ou de plus de 1450 gènes de Legionella pneumophila (cgMLST ad hoc). Une base de données commune européene est en cours de développement.
- L'analyse phylogénétique à partir de mapping sur un génome de référence ou d'assemblage de novo (construction d'un arbre phylogénétique et/ou analyse du nombre de SNP (Single Nucleotide Polymorphism) est l'analyse la plus discriminante. Elle est réalisée pour discriminer les grands clones de L. pneumophila (ST1, ST23,...) dans un contexte épidémique.
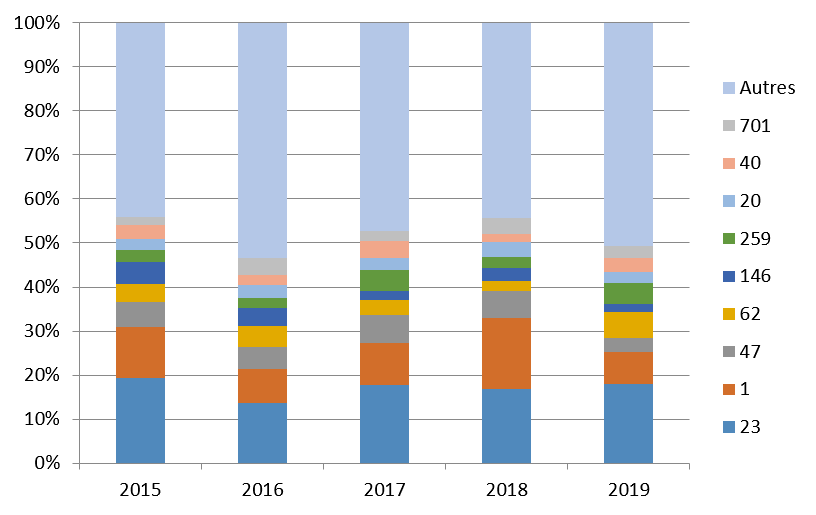
Les principaux ST retrouvés en France, ainsi que leur évolution dans le temps sont présentés dans la figure ci-dessous :
Figure. Evolution de la distribution des 9 principaux STs associés à l’infection en France de 2015 à 2019.
Méthode appliquée sur prélèvement : Nested-SBT
La technique de SBT peut être appliquée directement sur prélèvement clinique en s'affranchissant de l'isolement de souches. Cette méthode a été développée par le CNR. Le protocole est maintenant disponible sur le site de l’HPA (Health Public Agency).
Ginevra C, Lopez M, Forey F, Reyrolle M, Meugnier H, Vandenesch F, Etienne J, Jarraud S, Molmeret M. J Clin Microbiol. 2009;47:981-7.
Cette méthode doit être privilégiée secondairement à la mise en culture du prélèvement et après échec de celle-ci. Les prélèvements pulmonaires peuvent être adressés au CNR pour la réalisation de cette analyse.